Конвейер обработки данных представляет собой множество последовательных шагов, начиная от очистки необработанных данных и заканчивая построением оптимизированной модели машинного обучения для решения конкретных задач. Однако обработка данных — это тот самый этап, который требует наибольших усилий и времени, и который в дальнейшем определяет производительность моделей.
В этой статье сосредоточимся на том, как сделать отбор отдельных атрибутов (признаков) нашего набора данных, который является одной из основных задач фазы предварительной обработки. Но прежде чем погрузиться в кодирование и реализовать различные методы, используемые для подобных задач, давайте сначала определим, что подразумевается под отбором признаков. Отбор признаковએ — это процесс выбора подмножества атрибутов из набора данных, которые больше всего влияют на производительность модели, при этом не используются какие-либо преобразования.
Набор данных, на котором мы будем тренироваться, — это , опубликованный на Kaggle. Используя виртуальную машину ядра Kaggle, с ним можно работать напрямую, но лучше загрузить его на свой локальный компьютер.
Нам потребуется несколько сторонних библиотек Python и следующая команда позволит их установить:
$ pip3 install numpy pandas matplotlib sklearn
Предварительная обработка
Сначала загрузим набор данных в формат Dataframe с помощью pandasએ. Он состоит из 13 признаков (колонок, измерений, атрибутов) плюс метка и 270 строк.
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import MinMaxScaler
from sklearn.feature_selection import SelectKBest,chi2,RFE
from sklearn.ensemble import RandomForestClassifier
df = pd.read_csv("data/Heart_Disease_Prediction-ru.csv")
#df = pd.read_csv("data/Heart_Disease_Prediction.csv")
print('Размер Dataframe:', df.shape)
print(df.head(5))
Приведенный выше код импортирует необходимые библиотеки и считывает CSV‑файл набора данных из папки данных. Если вы скачали этот набор на свой компьютер, то Вам также необходимо создать отдельную папку данных. Путь к местоположению файла набора данных на своём компьютере можно изменить, соответствующим образом изменив код. Хочу отметить, что здесь для примеров используется не оригинальный файл Heart_Disease_Prediction.csv, а файл Heart_Disease_Prediction-ru.csv, где все категориальные значения переведены на русский язык.
После чего внимательно посмотрим на полученный Dataframe, вот результат:
Размер Dataframe: (270, 14) Возраст Пол ... Таллий Сердечное заболевание 0 70 1 ... 3 Есть 1 67 0 ... 7 Нет 2 57 1 ... 7 Есть 3 64 1 ... 7 Нет 4 74 0 ... 3 Нет [5 rows x 14 columns]
Получим обобщённую информацию о Dataframe, используя метод info():
print('\n\nИнформация о Dataframe df.info():')
print(df.info())
Здесь ясно видно, что пропуски значений отсутствуют. По всем признакам, во всех строках, все значения не‑нулевые non‑null и можно начать работу с нашим Dataframe, не беспокоясь об очистке пропусков.
Информация о Dataframe df.info():RangeIndex: 270 entries, 0 to 269 Data columns (total 14 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 Возраст 270 non-null int64 1 Пол 270 non-null int64 2 Тип боли в груди 270 non-null int64 3 BP 270 non-null int64 4 Холестерин 270 non-null int64 5 FBS свыше 120 270 non-null int64 6 Результаты EKG 270 non-null int64 7 Max HR 270 non-null int64 8 Стенокардия 270 non-null int64 9 ST депрессия 270 non-null float64 10 Наклон ST 270 non-null int64 11 Количество сосудов флюро 270 non-null int64 12 Таллий 270 non-null int64 13 Сердечное заболевание 270 non-null object dtypes: float64(1), int64(12), object(1)
Сохраним столбец метки в отдельной переменной и полностью удалим его (поэтому и используем inplace = True) из Dataframe. Этот шаг важен для разделения нашего набора данных на обучающий и проверочный в дополнение к тому, что необходима адаптация их к нашей модели.
label = df["Сердечное заболевание"]
df.drop("Сердечное заболевание", axis=1, inplace=True)
print('\n\nЗначение метки "Сердечное заболевание":')
print(label.value_counts())
Вот что получилось:
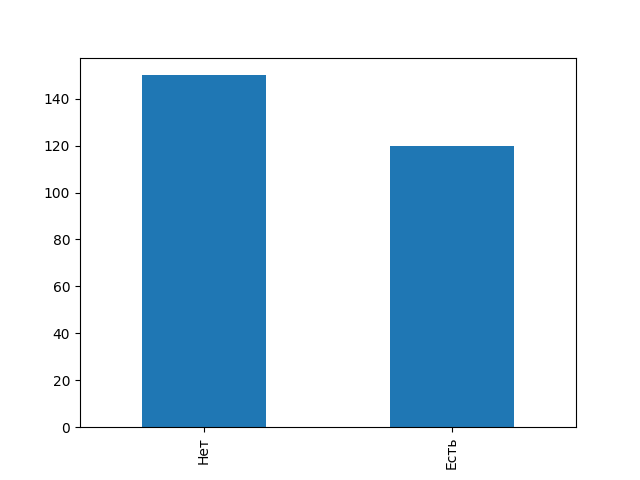
Значение метки "Сердечное заболевание": Нет 150 Есть 120 Name: Сердечное заболевание, dtype: int64
Важно всегда проверять, насколько сбалансирован наш набор. Большой дисбаланс между классами меньшинства и большинства отрицательно повлияет на модель в том смысле, что она будет наивно предсказывать только класс большинства. Однако, в нашем случае коэффициент дисбаланса, 150 / 120, составляет всего 1,25, что вполне себе удовлетворительно.
# создаем диаграмму, где ось x - это признак, а Y указывает значение метки label.value_counts().plot(kind="bar")
В нашем наборе данных есть признаки, имеющие категориальный характер. Однако, при печати этих столбцов мы наблюдаем, что они определяются, как данные целого типа и рассматриваются как целые числа, что может сбить с толку нашу модель, которая при обучении примет их за некий континиум значений, несмотря на то, что они дискретны по сути своей.
Типы данных признаков: Возраст int64 Пол int64 Тип боли в груди int64 BP int64 Холестерин int64 FBS свыше 120 int64 Результаты EKG int64 Max HR int64 Стенокардия int64 ST депрессия float64 Наклон ST int64 Количество сосудов флюро int64 Таллий int64 Сердечное заболевание object dtype: object
По этой причине явно изменим их тип на категориальный, используя метод astype() pandas.
categorical_features = ["Пол", "Тип боли в груди", "FBS свыше 120", "Результаты EKG", "Стенокардия", "Наклон ST", "Количество сосудов флюро", "Таллий"]
df[categorical_features] = df[categorical_features].astype("category")
Типы данных признаков: Возраст int64 Пол category Тип боли в груди category BP int64 Холестерин int64 FBS свыше 120 category Результаты EKG category Max HR int64 Стенокардия category ST депрессия float64 Наклон ST category Количество сосудов флюро category Таллий category dtype: object
Теперь будем масштабировать наши непрерывные функции с помощью MinMaxScaler. Это тип нормализации, когда значения загоняются в диапазоне от 0 до 1, согласно уравнению
X_{Norm} = \dfrac{(X - X_{Min})}{(X_{Max} - X_{Min})}.
continuous_features = set(df.columns) - set(categorical_features) scaler = MinMaxScaler() df_norm = df.copy() df_norm[list(continuous_features)] = scaler.fit_transform(df[list(continuous_features)])
Отбор признаков с помощью \chi^2
Тест \chi^2 (Распределение хи-квадратએ) используется в статистике для проверки независимости двух событий. Учитывая данные двух переменных, мы можем получить наблюдаемое число O и ожидаемое число E. \chi^2 измеряет, как ожидаемое число E и наблюдаемое число O отклоняются друг от друга.
При отборе признаков, поскольку \chi^2 проверяет степень независимости между двумя переменными, а мы хотим сохранить только признаки, наиболее зависимые от метки, то будем вычислять \chi^2 между каждым признаком и меткой, сохраняя только k признаки с наибольшими значениями. Будет правильно использовать SelectKBest и \chi^2 из модуля sklearn.feature_selection. При этом SelectKBest требует два гиперпараметра:
k: количество функций, которые мы хотим выбрать.score_func: функция, на которой основан процесс выбора.
X_new = SelectKBest(k=5, score_func=chi2).fit_transform(df_norm, label)
Отбор признаков с использованием рекурсивного исключения признаков (RFE)
В документации sklearn написано, что цель , — выбрать признаки путем рекурсивного рассмотрения все меньших и меньших наборов признаков. Будем использовать для этого модуль sklearn.feature_selection для импорта класса RFE. RFE требует двух гиперпараметров:
n_features_to_select: количество функций, которые мы хотим выбрать.estimator: какой тип модели машинного обучения будет использоваться для прогнозирования на каждой итерации при рекурсивном поиске соответствующего набора признаков.
rfe = RFE(estimator=RandomForestClassifier(), n_features_to_select=5) X_new = rfe.fit_transform(df_norm, label) X_new
Отбор признаков с использованием случайного леса (Random Forest)
Алгоритмы машинного обучения на основе деревьев (Random Forestએ), такие как DecisionTreeClassifier или их эквивалент для ансамблевого обучения RandomForestClassifier, используют набор деревьев, которые содержат узлы, полученные в результате разделения. Основная цель этих расщеплений — максимально возможно уменьшить количество «шумов» таких, как энтропия и коэффициент Джиниએ. Эти деревья, основанные на моделях могут рассчитать, насколько важен признак, рассчитав степень уменьшения «шумов» за счёт этого признака.
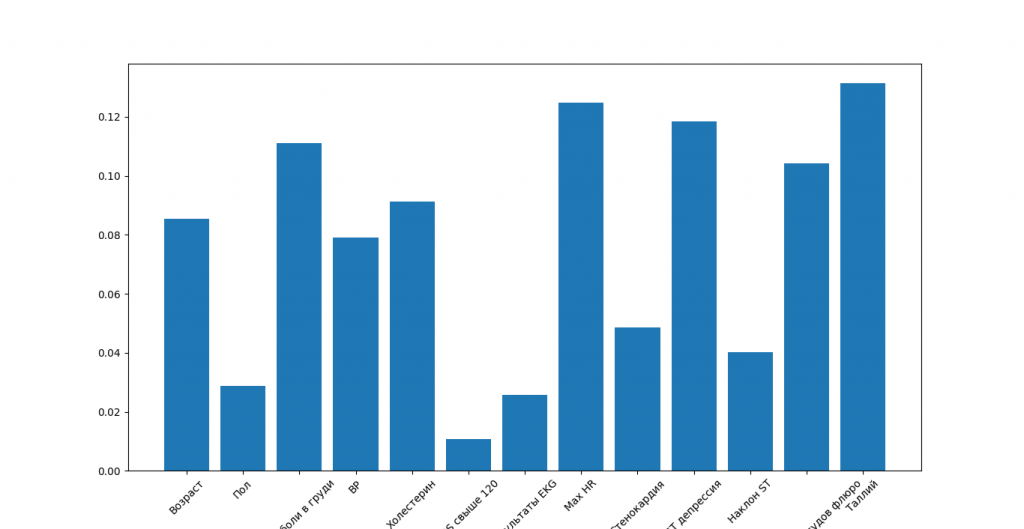
clf = RandomForestClassifier() clf.fit(df_norm, label) # create a figure to plot a bar, where x axis is features, and Y indicating the importance of each feature plt.figure(figsize=(12,12)) plt.bar(df_norm.columns, clf.feature_importances_) plt.xticks(rotation=45)
Гистограмма показывает важность каждого признака (атрибута, измерения, набора данных). В нашем случае Thallium и Number of vessels fluro являются наиболее важными характеристиками, но большинство из них имеют важное значение, и в этом случае в значительной степени стоит передать эти признаки нашей модели машинного обучения.
Теперь, когда выбраны наилучшие признаки, можно легко использовать любую модель классификатора sklearn, передать массив X_new и посмотреть, влияет ли это на точность модели с полным набором признаков.
В завершение соберём все, показанные здесь фрагменты, в единый полный код, который можно загрузить в IDLE Python:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import MinMaxScaler
from sklearn.feature_selection import SelectKBest,chi2,RFE
from sklearn.ensemble import RandomForestClassifier
df = pd.read_csv("data/Heart_Disease_Prediction-ru.csv")
#df = pd.read_csv("data/Heart_Disease_Prediction.csv")
print('Размер Dataframe:', df.shape)
print(df.head(5))
print('\n\nИнформация о Dataframe df.info():')
print(df.info())
# Используйте Dataframe.dtypes для
# определения данных в каждой колонке
datatypes = df.dtypes
print('\n\nТипы данных признаков:')
print(datatypes)
label = df["Сердечное заболевание"]
#label = df["Heart Disease"]
df.drop("Сердечное заболевание", axis=1, inplace=True)
print('\n\nЗначение метки "Сердечное заболевание":')
print(label.value_counts())
# создаем диаграмму, где ось x - это признак, а Y указывает значение метки
label.value_counts().plot(kind="bar")
categorical_features = ["Пол", "Тип боли в груди", "FBS свыше 120", "Результаты EKG", "Стенокардия", "Наклон ST", "Количество сосудов флюро", "Таллий"]
df[categorical_features] = df[categorical_features].astype("category")
# Используйте Dataframe.dtypes после категоризации
# определения данных в каждой колонке
datatypes = df.dtypes
print('\n\nТипы данных признаков:')
print(datatypes)
continuous_features = set(df.columns) - set(categorical_features)
scaler = MinMaxScaler()
df_norm = df.copy()
df_norm[list(continuous_features)] = scaler.fit_transform(df[list(continuous_features)])
X_new = SelectKBest(k=5, score_func=chi2).fit_transform(df_norm, label)
print('\n\nОтбор методом хи-квадрат\n', X_new)
rfe = RFE(estimator=RandomForestClassifier(), n_features_to_select=5)
X_new = rfe.fit_transform(df_norm, label)
print('\n\nОтбор методом случайных деревьев (Random Forest)\n', X_new)
clf = RandomForestClassifier()
clf.fit(df_norm, label)
# создаем диаграмму, где ось x - это признак, а Y указывает значимость каждого признака
plt.figure(figsize=(12,12))
plt.bar(df_norm.columns, clf.feature_importances_)
plt.xticks(rotation=45)
# рисуем всё сразу
plt.show()
Всё должно работать! Проверено — мин нет!
Заключение
Использование таких методов для выбора признаков варьируется от одной проблемы к другой, а также от одного признака к другому в зависимости от того, является ли их тип категориальным или непрерывным.
Кроме того, при выборе количества признаков я придерживаюсь итеративного подхода, пока k (в SelectKBest) не сойдется и производительность машинного обучения не слишком сильно увеличится.
В этой статье вы узнали, как:
- использовать \chi^2 для отбора признаков, сильно зависящих от метки;
- использовать RFE, чтобы рекурсивно найти оптимальный набор признаков с учетом оценки;
- использовать методы машинного обучения на основе дерева, такие как случайный лес, для отображения атрибутов, которые помогают максимально уменьшить искажения при разделении узлов.
Источник вдохновения:
![]() Отбор признаков с помощью Scikit-Learn в Python, опубликовано К ВВ, лицензия — Creative Commons Attribution-NonCommercial 4.0 International.
Отбор признаков с помощью Scikit-Learn в Python, опубликовано К ВВ, лицензия — Creative Commons Attribution-NonCommercial 4.0 International.
Респект и уважуха